“这意味着我们相较于第一代单细胞DNA测序技术(scDNA-seq)的重大进步。该新方法显著提高了实验通量,准确性和易用性。”德克萨斯大学安德森癌症中心Nicholas
Navin博士说到,“我们现在能够研究肿瘤细胞群中拷贝数的微小差异,而这在以前是不可能完成的。”1

在以往的研究中,受到可测序的肿瘤细胞数目限制,第一代单细胞DNA测序(scDNA-seq)技术噪音的影响,解析肿瘤扩展过程中的拷贝数进化问题非常困难。针对这一问题,Navin博士团队开发了一种具备单分子分辨率的单细胞DNA测序新方法。2
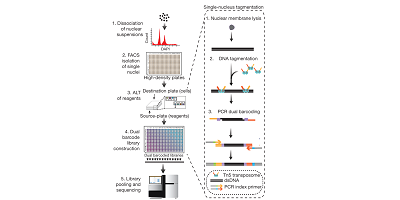
ACT(Acoustic Cell Tagmentation)声学细胞标记技术,从结合单核的荧光激活细胞分选(FACS)开始,分离单个细胞核。通过三步化学过程打断单细胞DNA,添加接头,获得带条形码的单细胞DNA文库。该化学过程采用贝克曼库尔特Echo声波移液技术3-4(ALT, acoustic liquid transfer)进行,利用声波能量快速、高效转移小体积液体。Echo以声波为能量隔空打物,无物理接触,不使用枪头,而移液体积可低至2.5 nL每液滴。

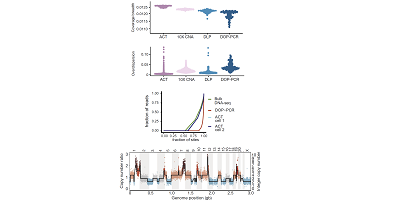
Navin团队比较了ACT技术、10X Genomics
CNV、DLP、DOP-PCR四种单细胞测序方法,ACT技术在测序覆盖度和技术噪音上表现出显著改善(P < 0.05)。而ACT技术(Gini系数,G = 0.728和0.678)的覆盖均匀性更接近bulk DNA-seq(G = 0.678),比DOP-PCR(G = 0.957)更均匀。采用ACT单细胞技术,研究人员观察到导管原位癌肿瘤的典型非整倍体细胞。
值得注意的是,与依赖全基因组放大的第一代scDNA-seq方法相比,Echo声波移液的ACT技术实验步骤更少,建库速度更快,仅仅3小时就可以完成。声波移液技术还表现出更高的实验通量,384孔板各孔加入不同index的耗时不超过4分钟。
Navin团队对来自8个人类三阴性乳腺癌样本和4种细胞系样本的16178个单细胞进行了测序及拷贝数分析,结果表明三阴性乳腺癌在最初的染色体不稳定性爆发后,基因拷贝数持续变化,并保持亚克隆多样性。目前,该团队正在调查其他癌症类型,探索这种癌症进化模型是否可以广泛应用到其他癌种。
参考文献:
1. https://www.mdanderson.org/newsroom/new-sequencing-approach-finds-triple-negative-breast-cancers-continue-accumulating-genetic-changes-during-tumor-growth.h00-159459267.html
2. Minussi,
D.C., Nicholson, M.D., Ye, H. et al. Breast tumours maintain a reservoir of
subclonal diversity during expansion. Nature 592, 302–308 (2021). https://doi.org/10.1038/s41586-021-03357-x
3.
US
Patent 6938987
4.
US
Patent 6938995
*本文涉及的内容与产品仅用于科研和工业,不用于临床诊断。